Ziel unserer Forschung ist es, ein tieferes Verständnis der molekularen Grundlagen und der klinischen Relevanz allergenspezifischer Antikörperantworten zu erlangen. Entsprechend dem Namen der Gruppe verfolgen wir einen synergetischen Ansatz, der Experimente im Labor und In-silico-Analysen kombiniert, um zum Fortschritt in der Diagnose und Behandlung von Allergien beizutragen.
Das Kennzeichen von Allergien vom Soforttyp ist die Bildung von Immunglobulin E (IgE)-Antikörpern. Ein seit langem diskutiertes Problem in der Allergiediagnostik ist die Tatsache, dass nicht alle Patienten, deren IgE an bestimmte Allergene bindet, allergische Reaktionen auf die jeweilige Allergenquelle zeigen. Es wurden mehrere Faktoren diskutiert, die die klinische Relevanz von allergenspezifischem IgE bestimmen, darunter die Konzentrationen allergenspezifischer Antikörper anderer Unterklassen wie IgG, die Bindungsstärke (Avidität) der IgE-Antikörper sowie die Anzahl und Verteilung der von IgE und IgG erkannten Bindungsstellen (Epitope).
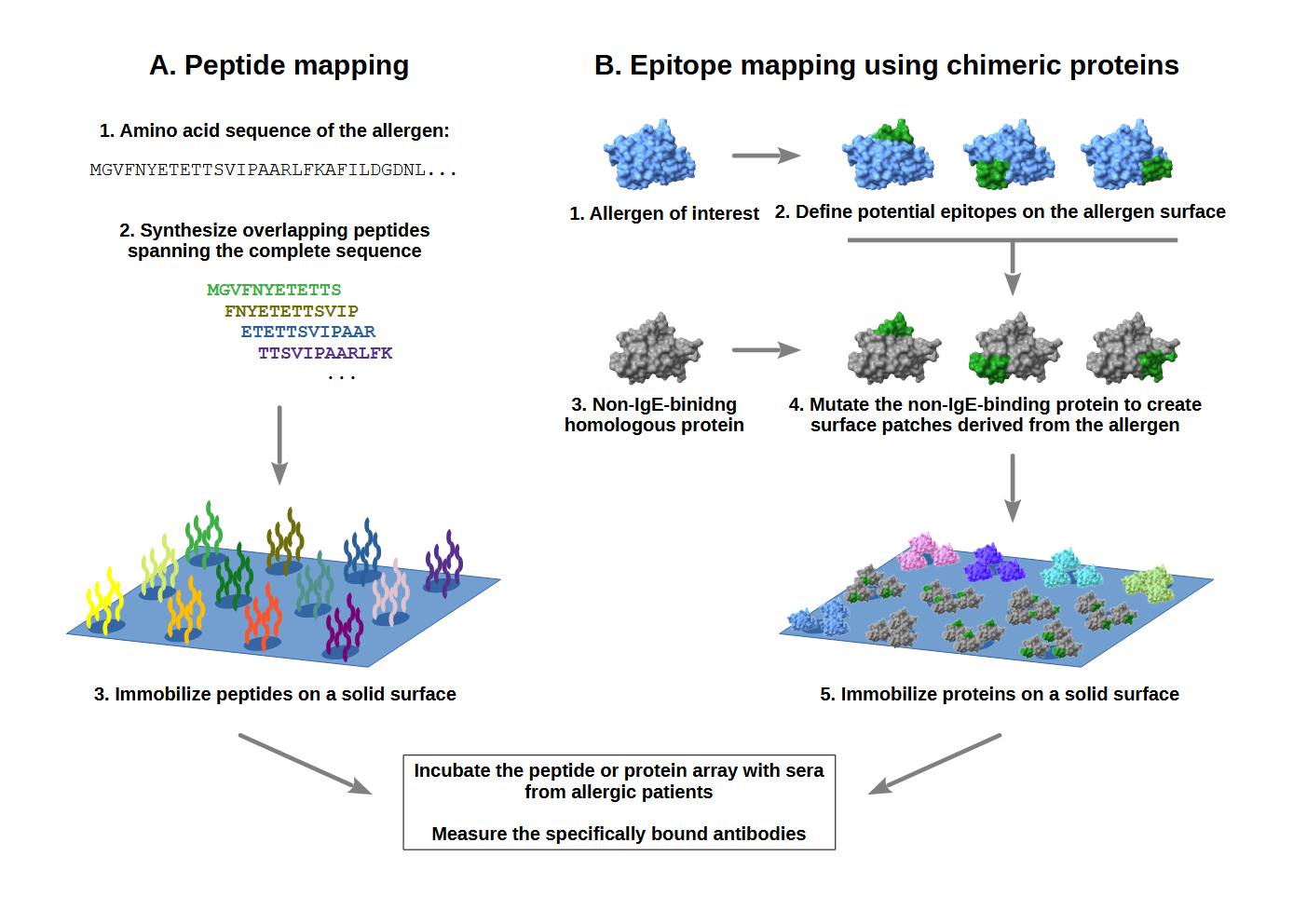
Epitope werden in der Regel identifiziert, indem Seren von Allergiepatienten mit einem Panel kurzer, sich überlappender synthetischer Peptide getestet werden, deren Sequenzen die vollständige Sequenz des Allergens abdecken (Abbildung 1, links). Atemwegsallergene kommen jedoch als intakte, gefaltete Proteine in Kontakt mit dem Immunsystem. Daher binden die meisten allergenspezifischen Antikörper an Epitope, die nur in den gefalteten Proteinen vorhanden sind und nicht durch kurze Peptide dargestellt werden können. Daher haben wir einen alternativen Ansatz entwickelt, der auf künstlichen chimären Proteinen basiert, die bestimmte, von der Oberfläche des Allergens abgeleitete Patches tragen (Abbildung 1, rechts).
Derzeit wenden wir diesen Ansatz an, um die Epitop-Bindungsmuster der allergenspezifischen Antikörper von Patienten zu charakterisieren, die gegen Birken- und Gräserpollen allergisch sind. Bei der Birkenpollenallergie wollen wir verstehen, warum die Patienten sehr unterschiedliche Muster allergischer Reaktionen auf bestimmte pflanzliche Nahrungsmittel wie Apfel, Haselnuss, Steinobst und Karotte zeigen, obwohl die IgE-Antikörper der meisten Patienten an Bet v 1, das Hauptallergen aus Birkenpollen, binden und mit Bet v 1 verwandten Allergenen in diesen Pflanzen kreuzreagieren. Bei der Gräserpollenallergie wollen wir die Entwicklung der Epitop-Erkennungsmuster der allergenspezifischen IgE- und IgG-Antikörper während des ersten Jahres der Allergenimmuntherapie (AIT) verfolgen. Dadurch erhoffen wir uns tiefere Einblicke in die immunologischen Mechanismen der AIT und die Ursachen für die unterschiedliche Wirksamkeit der Therapie.
Ausgewählte Publikationen
- Schmalz S, Mayr V, Shosherova A, Gepp B, Ackerbauer D, Sturm G, Bohle B, Breiteneder H, Radauer C (2022): Isotype-specific binding patterns of serum antibodies to multiple conformational epitopes of Bet v 1. J Allergy Clin Immunol 149: 1786-94. [Free full text]
- Tscheppe A, Palmberger D, van Rijt L, Kalic T, Mayr V, Palladino C, Kitzmüller C, Hemmer W, Hafner C, Bublin M, van Ree R, Grabherr R, Radauer C, Breiteneder H (2020): Development of a novel Ara h 2 hypoallergen with no IgE binding or anaphylactogenic activity. J Allergy Clin Immunol 145: 229-38. [Free full text]
Eines der ungelösten Probleme in der Allergieforschung ist die Frage, was ein Protein zu einem Allergen macht. Neben der Untersuchung des Verhaltens von Immunzellen oder Tiermodellen bei Kontakt mit allergenen und nicht-allergenen Proteinen ermöglicht es die Verfügbarkeit großer Allergendatenbanken in Kombination mit Sequenz-, Struktur- und Proteinfamiliendaten den Forschern nun, durch Sequenz- und Strukturanalyse neue Erkenntnisse zu gewinnen, die zu neuen experimentellen Ansätzen führen.
Wir versuchen, einige der folgenden Fragen zu beantworten. Wie ist die Verteilung allergener und nicht-allergener Proteine innerhalb allergenhaltiger Proteinfamilien und Superfamilien? Welche sequenzbezogenen Faktoren, wie z. B. Ähnlichkeit mit menschlichen Homologen, Ähnlichkeit mit Parasiten- oder Bakterienproteinen oder Sequenzkonservierung innerhalb einer Proteinfamilie, zeigen einen Zusammenhang mit Allergenität? Wie zuverlässig kann die IgE-Kreuzreaktivität zwischen homologen Allergenen auf der Grundlage von Sequenzähnlichkeitsdaten vorhergesagt werden?
Ausgewählte Publikationen
- Radauer C, Lackner P, Breiteneder H (2008): The Bet v 1 fold: an ancient, versatile scaffold for binding of large, hydrophobic ligands. BMC Evol Biol 8: 286. [Free full text]
Die meisten allergenen Proteine lassen sich in eine erstaunlich kleine Anzahl von Familien klassifizieren. Die Mitglieder dieser Proteinfamilien besitzen ähnliche Strukturen und in vielen Fällen auch ähnliche biochemische Funktionen. Dies führte zu der Hypothese, dass die Allergenität von strukturellen oder funktionellen Merkmalen der Proteine abhängt. Darüber hinaus ist die Zugehörigkeit zu einer gemeinsamen Proteinfamilie eine Voraussetzung für die Kreuzreaktivität von IgE-Antikörpern und T-Zellen.
Um Wissenschafter:innen und Kliniker:innen zu helfen, sich einen Überblick über Allergenfamilien und ihre allergenen Mitglieder zu verschaffen, haben wir die AllFam-Allergenfamiliendatenbank entwickelt, eine Webressource, die es dem Benutzer ermöglicht, Proteinfamiliendaten für Allergenen zu erhalten, die aus der WHO/IUIS-Allergen-Nomenklaturdatenbank und AllergenOnline stammen.
Ausgewählte Publikationen
- Radauer C, Breiteneder H (2019): Allergen databases - A critical evaluation. Allergy 74: 2057-2060. [Free full text]
- Radauer C, Bublin M, Wagner S, Mari A, Breiteneder H (2008): Allergens are distributed into few protein families and possess a restricted number of biochemical functions. J Allergy Clin Immunol 121: 847-52. [Full text]

Christian Radauer
GruppenleiterAndrea Martschini
PhD-StudentinTel.: +43 1 40400 51170
E-Mail: andrea.martschini (at) meduniwien.ac.at
Petra Natascha Hendler
Administrative AssistentinAlumni
- Stefanie Ottersbach (PhD-Studentin 2018-22)
- Vanessa Mayr (Biomedizinische Analytikerin 2019-21)
- Gerlinde Hofstetter (Biomedizinische Analytikerin 2010-14)
- Eva Rauch-Guhsl (PhD-Studentin 2010-14)
Christian Radauer: Curriculum Vitae
Christian Radauer studierte Biochemie an der Universität Wien. Sein Doktoratsstudium absolvierte er in der Gruppe von Heimo Breiteneder am ehemaligen Institut für Allgemeine und Experimentelle Pathologie der Universität Wien (heute Institut für Pathophysiologie und Allergieforschung der Medizinischen Universität Wien). Christian Radauer erhielt seinen Doktortitel im Jahr 2000. Er setzte seine Karriere im Labor von Heimo Breiteneder als Post-Doc fort. Während eines kurzen Forschungsaufenthalts in der Gruppe von Peter Lackner am Institut für Molekularbiologie der Universität Salzburg sammelte er zusätzliche Erfahrungen in der Bioinformatik. Im Jahr 2009 habilitierte er sich für das Fach Molekulare Allergologie.
Christian Radauers Forschungsinteressen konzentrieren sich auf den Bereich der molekularen Allergologie. Themen sind die Aufklärung der molekularen Eigenschaften von Allergien, die Faktoren, die bestimmte Proteine allergen machen, die molekulare Grundlage der Kreuzreaktivität und die klinische Relevanz von patientenspezifischen Repertoires allergenspezifischer Immunglobuline. Seit April 2022 ist Christian Radauer wissenschaftlicher Ko-Vorsitzender des WHO/IUIS-Unterausschusses für Allergen-Nomenklatur. Die Aufgabe dieses Komitees ist es, eine offizielle, eindeutige Nomenklatur für allergene Proteine zu entwickeln und die offiziellen Allergennamen in der WHO/IUIS Allergen Nomenclature Database der wissenschaftlichen Gemeinschaft zugänglich zu machen.